Журнал «Здоровье ребенка» 8 (68) 2015
Вернуться к номеру
Транзиторный неонатальный сахарный диабет, ассоциированный с нарушением импринтинга хромосомы 6q24. Часть 1. Импринтированный кластер PLAGL1/HYMAI
Авторы: Абатуров А.Е., Агафонова Е.А., Бабич В.Л. - ГУ «Днепропетровская медицинская академия Министерства здравоохранения Украины»
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
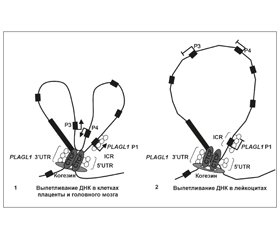
В статье приведены современные данные о механизмах генетических нарушений, приводящих к развитию неонатального сахарного диабета, ассоциированного с нарушением импринтинга хромосомы 6q24. Статья содержит данные о строении импринтированного кластера PLAGL1/HYMAI, гена PLAGL1 и протеина ZAC1, некодирующих РНК HYMAI и ZAC-AS, дифференциально метилированной области кластера PLAGL1/HYMAI. Показано, что гиперметилирование ICR кластера PLAGL1/HYMAI сопровождается транскрипционным молчанием гена PLAGL1 в клетках различных злокачественных опухолей человека, в том числе рака яичников, колоректального рака, саркомы, глиобластомы и рабдомиосаркомы, а гипометилирование — транзиторным сахарным диабетом новорожденных.
У статті наведені сучасні дані про механізми генетичних порушень, що призводять до розвитку неонатального цукрового діабету, асоційованого з порушенням імпринтингу хромосоми 6q24. Стаття містить дані про будову імпринтованого кластера PLAGL1/HYMAI, гена PLAGL1 і протеїну ZAC1, некодуючих РНК HYMAI і ZAC-AS, диференційно метильованої ділянки кластера PLAGL1/HYMAI. Показано, що гіперметилювання ICR кластера PLAGL1/HYMAI супроводжується транскрипційним мовчанням гена PLAGL1 у клітинах різних злоякісних пухлин людини, у тому числі раку яєчників, колоректального раку, саркоми, гліобластоми та рабдоміосаркоми, а гіпометилювання — транзиторним цукровим діабетом новонароджених.
The article presents modern data on the mechanisms of genetic disorders that lead to the development of neonatal diabetes mellitus associated with the violation of imprinting of 6q24 chromosome. This article contains information about the structure of the imprinted cluster PLAGL1/HYMAI, gene and protein PLAGL1 ZAC1, non-coding RNAs and HYMAI ZAC-AS, the differentially methylated area PLAGL1/HYMAI cluster. It is shown that the cluster ICR PLAGL1/HYMAI hypermethylation is accompanied with PLAGL1 gene transcriptional silencing in cells of various human malignancies, including ovarian cancer, colorectal cancer, sarcoma, glioblastoma and rhabdomyosarcoma, and hypomethylation is associated with transient neonatal diabetes mellitus.
импринтированный кластер PLAGL1/HYMAI.
імпринтований кластер PLAGL1/HYMAI.
cluster of imprinted PLAGL1/HYMAI.
Статья опубликована на с. 56-61
Импринтированный кластер
PLAGL1/HYMAI
Регуляция импринтированной экспрессии
Модификация гистоновых белков
1. Diatloff-Zito C. Genetic and epigenetic defects at the 6q24 imprinted locus in a cohort of 13 patients with transient neonatal diabetes: new hypothesis raised by the finding of a unique case with hemizygotic deletion in the critical region / C. Diatloff-Zito, A. Nicole, G. Marcelin et al. // J. Med. Genet. — 2007 Jan. — 44(1). — 31-7. — doi: 10.1136/jmg.2006.044404.
2. Kamiya M. The cell cycle control gene ZAC/PLAGL1 is imprinted — a strong candidate gene for transient neonatal diabetes / Kamiya M., Judson H., Okazaki Y. et al. // Hum. Mol. Genet. — 2000, Feb 12. — 9(3). — 453-60. — doi: 10.1093/hmg/9.3.453.
3. Iglesias-Platas I. Altered expression of the imprinted transcription factor PLAGL1 deregulates a network of genes in the human IUGR placenta / I. Iglesias-Platas, A. Martin-Trujillo, P. Petazzi et al. // Hum. Mol. Genet. — 2014, Dec 1. — 23(23). — 6275-85. — doi: 10.1093/hmg/d.
4. Arima T. A novel imprinted gene, HYMAI, is located within an imprinted domain on human chromosome 6 containing ZAC / T. Arima, R.A. Drewell, M. Oshimura, N. Wake, M.A. Surani // Genomics. — 2000, Aug 1. — 67(3). — 248-55. — doi: 10.1006/geno.2000.6266.
5. Varrault A. Zac1 regulates an imprinted gene network critically involved in the control of embryonic growth / A. Varrault, C. Gueydan, A. Delalbre et al. // Dev. Cell. — 2006 Nov. — 11(5). — 711-22. — doi: 10.1002/dvdy.20373.
6. Iglesias-Platas I. Imprinting at the PLAGL1 domain is contained within a 70-kb CTCF/cohesin-mediated non-allelic chromatin loop / I. Iglesias-Platas, F. Court, C. Camprubi et al. // Nucleic. Acids Res. — 2013, Feb 1. — 41(4). — 2171-9. — doi: 10.1093/nar/gks1355.
7. Abdollahi A. LOT1 (PLAGL1/ZAC1), the candidate tumor suppressor gene at chromosome 6q24-25, is epigenetically regulated in cancer / A. Abdollahi, D. Pisarcik, D. Roberts et al. // J. Biol. Chem. — 2003, Feb 21. — 278(8). — 6041-9. — doi: 10.1074/jbc.M210361200.
8. Abdollahi A. LOT1 (ZAC1/PLAGL1) and its family members: mechanisms and functions // J. Cell. Physiol. — 2007. — 210. — 16-25. — doi: 10.1002/jcp.20835.
9. Mackay D.J. Relaxation of imprinted expression of ZAC and HYMAI in a patient with transient neonatal diabetes mellitus D.J. Mackay, A.M. Coupe, J.P. Shield et al. // Hum. Genet. — 2002 Feb. — 110(2). — 139-44. — doi: 10.1007/s00439-001-0671-5.
10. Hoffmann A., Spengler D. Transient neonatal diabetes mellitus gene Zac1 impairs insulin secretion in mice through Rasgrf1 // Mol. Cell. Biol. — 2012 Jul. — 32(13). — 2549-60. — doi: 10.1128/MCB.06637-11.
11. Smith R.J. The mouse Zac1 locus: basis for imprinting and comparison with human ZAC/ R.J. Smith, P. Arnaud, G. Konfortova et al. // Gene. — 2002, Jun 12. — 292(1–2). — 101-12. — doi: 10.1016/S0378-1119(02)00666-2.
12. Valleley E.M., Cordery S.F., Bonthron D.T. Tissue-specific imprinting of the ZAC/PLAGL1 tumour suppressor gene results from variable utilization of monoallelic and biallelic promoters // Hum. Mol. Genet. — 2007, Apr 15. — 16(8). — 972-81. — doi: 10.1093/hmg/ddm041.
13. Chotalia M. Transcription is required for establishment of germline methylation marks at imprinted genes / M. Chotalia, S.A. Smallwood, N. Ruf et al. // Genes. Dev. — 2009, Jan 1. — 23(1). — 105-17. — doi: 10.1101/gad.495809.
14. Hoffmann A. Transcriptional activities of the zinc finger protein Zac are differentially controlled by DNA binding / A. Hoffmann, E. Ciani, J. Boeckardt et al. // Mol. Cell. Biol. — 2003 Feb. — 23(3). — 988-1003. — doi: 10.1128/MCB.23.3.988-1003.2003.
15. Godlewski J. PLAGL1 protein is differentially expressed in the nephron segments and collecting ducts in human kidney / J. Godlewski, B.E. Krazinski, J. Kiezun et al. // Folia Histochem. Cytobiol. — 2015. — 53(1). — 96-104. — doi: 10.5603/FHC.a2015.0011.
16. Varrault A. hZAC encodes a zinc finger protein with antiproli–ferative properties and maps to a chromosomal region frequently lost in cancer / A. Varrault, E. Ciani, F. Apiou et al. // Proc. Natl Acad. Sci. USA. — 1998. — 95. — 8835-8840. — doi: 10.1073/pnas.95.15.8835.
17. Ahrén B. Role of pituitary adenylate cyclase-activating polypeptide in the pancreatic endocrine system / Ann. N. Y. Acad. Sci. — 2008 Nov. — 1144. — 28-35. — doi: 10.1196/annals.1418.003.
18. Ho C.L. Modulation of the Zac1's transactivation and coactivation functions via PML and Daxx within distinct subcellular loca–lizations / C.L. Ho, Y.C. Huang, C.K. Tai et al. // Int. J. Biochem. Cell. Biol. — 2010 Jun. — 42(6). — 902-10. — doi: 10.1016/j.biocel.2010.01.020.
19. Sakurai Y. Trophic effects of PACAP on pancreatic islets: a mini-review/ Y. Sakurai, N. Shintani, A. Hayata, H. Hashimoto, A. Baba // J. Mol. Neurosci. — 2011 Jan. — 43(1). — 3-7. — doi: 10.1007/s12031-010-9424-z.
20. Rozenfeld-Granot G. A positive feedback mechanism in the transcriptional activation of Apaf-1 by p53 and the coactivator –Zac-1 / G. Rozenfeld-Granot, J. Krishnamurthy, K. Kannan et al. // Oncogene. — 2002, Feb 28. — 21(10). — 1469-76. doi: 10.1038/sj/onc/1205218.
21. Huang S.M., Stallcup M.R. Mouse Zac1, a transcriptional coactivator and repressor for nuclear receptors // Mol. Cell. Biol. — 2000 Mar. — 20(5). — 1855-67. PMCID: PMC85366.
22. Daniel G. Imprinted Zac1 in neural stem cells/ G. Daniel, U. Schmidt-Edelkraut, D. Spengler, A. Hoffmann // World J. Stem. Cells. — 2015, Mar 26. — 7(2). — 300-14. — doi: 10.4252/wjsc.v7.i2.300.
23. Iglesias-Platas I. Characterization of novel paternal ncRNAs at the Plagl1 locus, including Hymai, predicted to interact with regulators of active chromatin / I. Iglesias-Platas, A. Martin-Trujillo, D. Cirillo et al. // PLoS One. — 2012. — 7(6). — e38907. — doi: 10.1371/journal.pone.0038907.
24. Arima T. A conserved imprinting control region at the HYMAI/ZAC domain is implicated in transient neonatal diabetes mellitus / T. Arima, R.A. Drewell, K.L. Arney et al. // Hum. Mol. Genet. — 2001, Jul 1. — 10(14). — 1475-83. — doi: 10.1093/hmg/10.14.1475.
25. Varrault A. Characterization of the methylation-sensitive promoter of the imprinted ZAC gene supports its role in transient neonatal diabetes mellitus / A. Varrault, B. Bilanges, D.J. Mackay et al. // J. Biol. Chem. — 2001, Jun 1. — 276(22). — 18653-6. — doi: 10.1074/jbc.C100095200.
26. Cheong C.Y. Germline and somatic imprinting in the nonhuman primate highlights species differences in oocyte methylation / C.Y. Cheong, K. Chng, S. Ng et al. // Genome Res. — 2015 May. — 25(5). — 611-23. — doi: 10.1101/gr.183301.114.
27. Peille A.L. Prognostic value of PLAGL1-specific CpG site methylation in soft-tissue sarcomas / A.L. Peille, V. Brouste, A. Kauffmann et al. // PLoS One. — 2013, Nov 15. — 8(11). — e80741. — doi: 10.1371/journal.pone.0080741.
28. Yin F. Tumor suppressor genes associated with drug resistance in ovarian cancer (review) / F. Yin, X. Liu, D. Li et al. // Oncol. Rep. — 2013 Jul. — 30(1). — 3-10. — doi: 10.3892/or.2013.2446.
29. Kowalczyk A.E. Altered expression of the PLAGL1 (ZAC1/LOT1) gene in colorectal cancer: Correlations to the clinicopathological parameters / A.E. Kowalczyk, B.E. Krazinski, J. Godlewski et al. // Int. J. Oncol. — 2015 Sep. — 47(3). — 951-62. — doi: 10.3892/ijo.2015.3067.
30. Hide T. Sox11 prevents tumorigenesis of glioma-initiating cells by inducing neuronal differentiation / T. Hide, T. Takezaki, Y. Nakatani et al. // Cancer. Res. — 2009, Oct 15. — 69(20). — 7953-9. — doi: 10.1158/0008-5472.CAN-09-2006.
31. Rezvani G. A set of imprinted genes required for normal body growth also promotes growth of rhabdomyosarcoma cells / G. Rezvani, J.C. Lui, K.M. Barnes, J. Baron // Pediatr. Res. — 2012 Jan. — 71(1). — 32-8. — doi: 10.1038/pr.2011.6.
32. Mackay D.J., Temple I.K. Transient neonatal diabetes mellitus type 1 // Am. J. Med. Genet. C Semin. Med. Genet. — 2010, Aug 15. — 154C(3). — 335-42. — doi: 10.1002/ajmg.c.30272.
1. Diatloff-Zito C et al. Genetic and epigenetic defects at the 6q24 imprinted locus in a cohort of 13 patients with transient neonatal diabetes: new hypothesis raised by the finding of a unique case with hemizygotic deletion in the critical region. C. J Med Genet. 2007 Jan;44(1):31-7. doi: 10.1136/jmg.2006.044404.
2. Kamiya M et al. The cell cycle control gene ZAC/PLAGL1 is imprinted--a strong candidate gene for transient neonatal diabetes. Hum Mol Genet. 2000 Feb 12;9(3):453-60. doi: 10.1093/hmg/9.3.453
3. Iglesias-Platas I et al. Altered expression of the imprinted transcription factor PLAGL1 deregulates a network of genes in the human IUGR placenta. Hum Mol Genet. 2014 Dec 1;23(23):6275-85. doi: 10.1093/hmg/d
4. Arima T et al. A novel imprinted gene, HYMAI, is located within an imprinted domain on human chromosome 6 containing ZAC. Genomics. 2000 Aug 1;67(3):248-55. doi: 10.1006/geno.2000.6266
5. Varrault A et al. Zac1 regulates an imprinted gene network critically involved in the control of embryonic growth. Dev Cell. 2006 Nov;11(5):711-22. doi: 10.1002/dvdy.20373.
6. Iglesias-Platas I et al. Imprinting at the PLAGL1 domain is contained within a 70-kb CTCF/cohesin-mediated non-allelic chromatin loop. Nucleic Acids Res. 2013 Feb 1;41(4):2171-9. doi: 10.1093/nar/gks1355.
7. Abdollahi A et al. LOT1 (PLAGL1/ZAC1), the candidate tumor suppressor gene at chromosome 6q24-25, is epigenetically regulated in cancer. J Biol Chem. 2003 Feb 21;278(8):6041-9., doi: 10.1074/jbc.M210361200
8. Abdollai A. LOT1 (ZAC1/PLAGL1) and its family members: mechanisms and functions. J Cell Physiol. 2007;210:16–25. doi: 10.1002/jcp.20835.
9. Mackay DJ et al. Relaxation of imprinted expression of ZAC and HYMAI in a patient with transient neonatal diabetes mellitus. Hum Genet. 2002 Feb;110(2):139-44. doi: 10.1007/s00439-001-0671-5.
10. Hoffmann A, Spengler D. Transient neonatal diabetes mellitus gene Zac1 impairs insulin secretion in mice through Rasgrf1. Mol Cell Biol. 2012 Jul;32(13):2549-60. doi: 10.1128/MCB.06637-11.
11. Smith RJ et al. The mouse Zac1 locus: basis for imprinting and comparison with human ZAC. Gene. 2002 Jun 12;292(1-2):101-12. doi:10.1016/S0378-1119(02)00666-2.
12. Valleley EM, Cordery SF, Bonthron T. Tissue-specific imprinting of the ZAC/PLAGL1 tumour suppressor gene results from variable utilization of monoallelic and biallelic promoters. Hum Mol Genet. 2007 Apr 15;16(8):972-81. doi: 10.1093/hmg/ddm041
13. Chotalia M et al.Transcription is required for establishment of germline methylation marks at imprinted genes. Genes Dev. 2009 Jan 1;23(1):105-17. doi: 10.1101/gad.495809.
14. Hoffmann A et al. Transcriptional activities of the zinc finger protein Zac are differentially controlled by DNA binding. Mol Cell Biol. 2003 Feb;23(3):988-1003. doi: 10.1128/MCB.23.3.988-1003.2003.
15. Godlewski J t al. PLAGL1 protein is differentially expressed in the nephron segments and collecting ducts in human kidney. Folia Histochem Cytobiol. 2015;53(1):96-104. doi: 10.5603/FHC.a2015.0011
16. Varrault A et al. hZAC encodes a zinc finger protein with antiproliferative properties and maps to a chromosomal region frequently lost in cancer. Proc Natl Acad Sci USA. 1998;95:8835–8840. doi: 10.1073/pnas.95.15.8835.
17. Ahrén B. Role of pituitary adenylate cyclase-activating polypeptide in the pancreatic endocrine system/ Ann N Y Acad Sci. 2008 Nov;1144:28-35. doi: 10.1196/annals.1418.003.
18. Ho CL et al. Modulation of the Zac1's transactivation and coactivation functions via PML and Daxx within distinct subcellular localizations. Int J Biochem Cell Biol. 2010 Jun;42(6):902-10. doi: 10.1016/j.biocel.2010.01.020.
19. Sakurai Y. Trophic effects of PACAP on pancreatic islets: a mini-review/ Y. Sakurai, N. Shintani, A. Hayata, H. Hashimoto, A. Baba. J Mol Neurosci. 2011 Jan;43(1):3-7. doi: 10.1007/s12031-010-9424-z.
20. Rozenfeld-Granot G et al. A positive feedback mechanism in the transcriptional activation of Apaf-1 by p53 and the coactivator Zac-1. Oncogene. 2002 Feb 28;21(10):1469-76. doi: 10.1038/sj/onc/1205218.
21. Huang SM, Stallcup MR. Mouse Zac1, a transcriptional coactivator and repressor for nuclear receptors. Mol Cell Biol. 2000 Mar;20(5):1855-67. PMCID: PMC85366
22. Daniel G. Imprinted Zac1 in neural stem cells/ G. Daniel, U. Schmidt-Edelkraut, D. Spengler, A. Hoffmann. World J Stem Cells. 2015 Mar 26;7(2):300-14. doi: 10.4252/wjsc.v7.i2.300.
23. Iglesias-Platas I et al. Characterization of novel paternal ncRNAs at the Plagl1 locus, including Hymai, predicted to interact with regulators of active chromatin. PLoS One. 2012;7(6):e38907. doi: 10.1371/journal.pone.0038907.
24. Arima T et al. A conserved imprinting control region at the HYMAI/ZAC domain is implicated in transient neonatal diabetes mellitus. Hum Mol Genet. 2001 Jul 1;10(14):1475-83. doi: 10.1093/hmg/10.14.1475.
25. Varrault A et al. Characterization of the methylation-sensitive promoter of the imprinted ZAC gene supports its role in transient neonatal diabetes mellitus. J Biol Chem. 2001 Jun 1;276(22):18653-6. doi: 10.1074/jbc.C100095200.
26. Cheong CY et al. Germline and somatic imprinting in the nonhuman primate highlights species differences in oocyte methylation. Genome Res. 2015 May;25(5):611-23. doi: 10.1101/gr.183301.114.
27. Peille AL et al. Prognostic value of PLAGL1-specific CpG site methylation in soft-tissue sarcomas. PLoS One. 2013 Nov 15;8(11):e80741. doi: 10.1371/journal.pone.0080741.
28. Yin F et al. Tumor suppressor genes associated with drug resistance in ovarian cancer (review). Oncol Rep. 2013 Jul;30(1):3-10. doi: 10.3892/or.2013.2446.
29. Kowalczyk AE et al. Altered expression of the PLAGL1 (ZAC1/LOT1) gene in colorectal cancer: Correlations to the clinicopathological parameters. Int J Oncol. 2015 Sep;47(3):951-62. doi: 10.3892/ijo.2015.3067.
30. Hide T et al. Sox11 prevents tumorigenesis of glioma-initiating cells by inducing neuronal differentiation. Cancer Res. 2009 Oct 15;69(20):7953-9. doi: 10.1158/0008-5472.CAN-09-2006.
31. Rezvani G et al. A set of imprinted genes required for normal body growth also promotes growth of rhabdomyosarcoma cells. Pediatr Res. 2012 Jan;71(1):32-8. doi: 10.1038/pr.2011.6.
32. Mackay DJ, Temple IK. Transient neonatal diabetes mellitus type 1. Am J Med Genet C Semin Med Genet. 2010 Aug 15;154C(3):335-42. doi: 10.1002/ajmg.c.30272

/57.jpg)
/58.jpg)
/60.jpg)